遺伝子変異情報から新型コロナウイルス変異株の感染力を予測できる数理モデルを開発
バイオインフォマティクス部門|2023.3.23
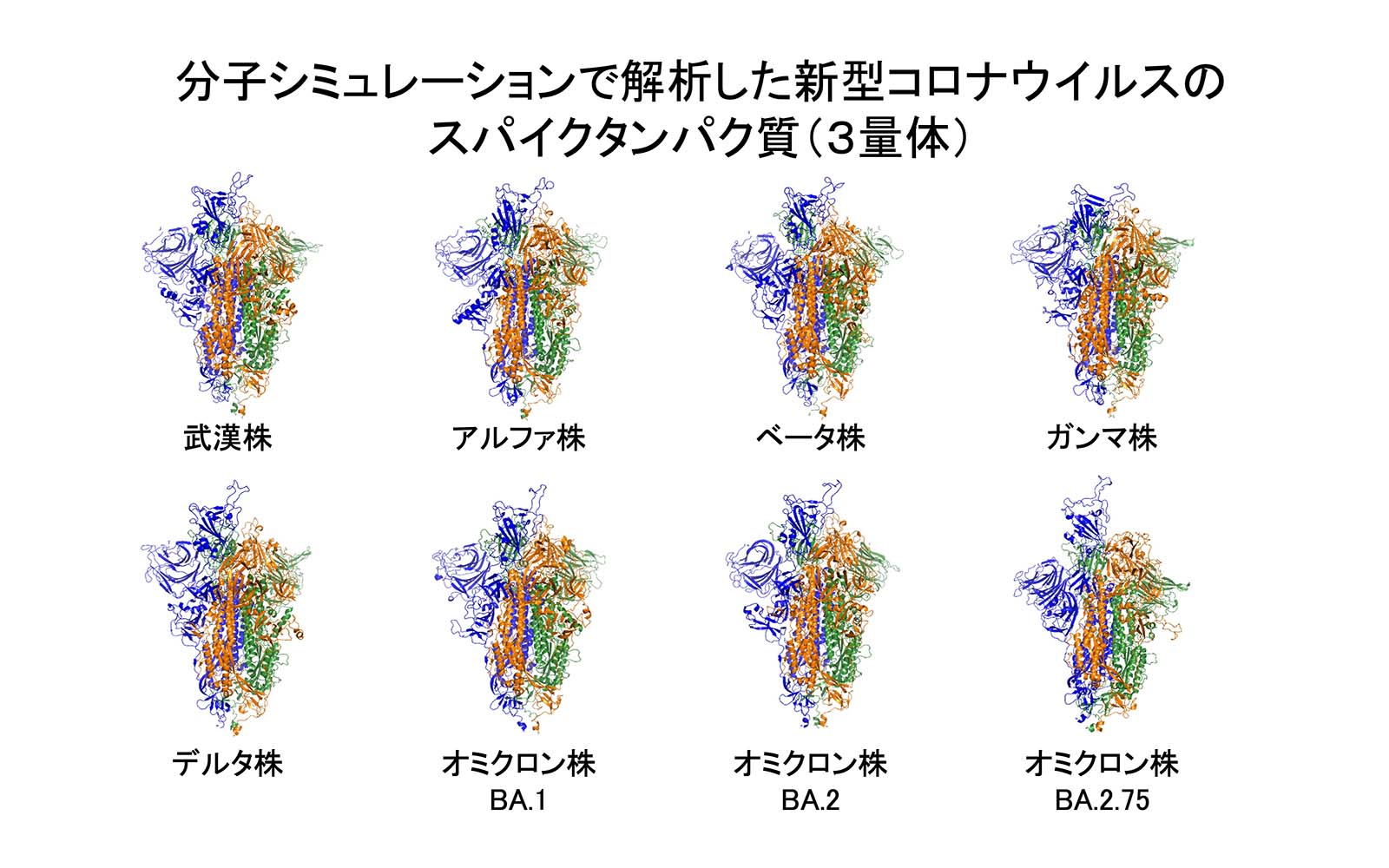
新型コロナウイルススパイクタンパク質
高岡裕教授らの研究チームは、分子シミュレーション解析と 数理モデルによって、各種コロナウイルス変異株の細胞への 感染性予測を可能にしました。まず、新型コロナ6変異株(武漢, アルファ, ベータ, ガンマ, デルタ, オミクロン BA.1, オミクロン BA.2)の感染力1:1.5:1.5:2:2:4:6を再現すべく、6種類のスパイクタンパク質の立体構造をホモロジーモデリング法で解析しました。その後、ヒトACE2タンパク質とのドッキング解析に供し、そのドッキング解析結果を用いて、新型コロナの感染力を遺伝子変異情報から予測できる数理モデルを導出しました。その結果、高精度で感染力を予測できるようになりました(1)。
これらの研究結果を応用することで、新規変異株が出現しても、すぐに感染性予測を発出できるようになりました(2)。これらの手法を、感染症薬をはじめとする創薬に活かすべく、今後も研究に取り組んでまいります。
論文
(1)Microbial Risk Analysis 22:100227, doi:10.1016/j.mran.2022.100227, 2022
(2)Microorganisms 10(10):2090, doi:10.3390/microorganisms10102090, 2022